Les bactéries constituent un important sujet d'étude dans le secteur biomédical car elles causent de nombreuses infections du corps humain. Les méthodes de diagnostic actuelles basées principalement sur la culture de populations en boite de Pétri permettent d'identifier la bactérie responsable de l'infection et l'antibiotique qui la stoppera en 48 heures. Afin de réduire ce temps de diagnostic nous proposons de travailler sur l’analyse de bactérie à l’échelle de la cellule unique en exploitant les forces optiques.
Ces dernières sont généralement utilisées en espace libre à travers un microscope, on parle alors de pinces optiques. Cependant, d'autres dispositifs optofluidiques ont récemment émergé, basés sur l'utilisation de composants optiques intégrés sur puce tels que des cristaux photoniques et microcavités. Les faibles puissances mises en jeu dans ces nouveaux dispositifs sont particulièrement adaptées à la manipulation d’objets biologiques.
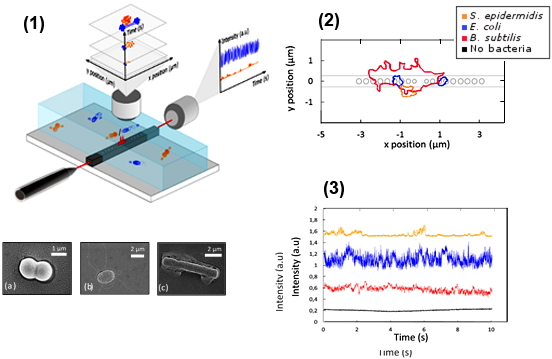
Figure : (1) Configuration de piégeage et images SEM de la bactérie étudiée : (a) diplocoque de
S. epidermidis, (b)
E. coli et (c) agrégat de
B. subtilis ; (2) forme globale des trajectoires des bactéries piégées sur la microcavité SOI ; (3) Exemple de l'intensité transmise par la microcavité pendant 10 secondes de piégeage pour le diplocoque diplocoque de S. epidermidis (orange),
E. coli (bleu), agrégat de
B. subtilis (rouge) et sans bactérie piégée (dans les bactéries et en milieu aqueux) (noir).
Nous avons développé un dispositif de piégeage optique basé sur l’utilisation de microcavités SOI et dédié à l’étude des bactéries. L'analyse spatiale du mouvement brownien d’une bactérie dans le piège, corrélée au signal transmis par la structure optique permet une caractérisation précise du comportement de l'objet piégé. De plus, l’analyse des fluctuations du signal transmis en fonction du temps pendant le piégeage, permet de mettre en évidence la signature de chaque type de bactérie piégée. Grace à cette méthode, nous avons démontré le piégeage et l’identification de trois bactéries :
S. epidermidis,
E. coli et
B. subtilis. Nous souhaitons étendre cette étude des interactions bactérie/champ électromagnétique à la discrimination de différents genres ou espèces bactériennes. D’autre part, nous envisageons de corréler la signature optique d'une bactérie piégée en réponse à des événements externes (vie et mort, division cellulaire).
Les microrésonateurs optiques utilisés dans ce projet sont fabriquées à la PTA par J-B. Jager.